降维技术的好处:
1.使得数据集更易使用
2.降低很多算法的计算开销
3.取出噪声
4.使得结果易懂
在已标注和未标注的数据上都有降维技术,降维的方法:
1.主成分分析(Principal Component Analysis,PCA)。在PCA中,数据从原来的坐标系转换到新的坐标系,新坐标系的选择是由数据本身决定的。第一个新坐标轴选择的是原始数据中方差最大的方向,第二个新坐标轴的选择和第一个坐标轴正交且具有最大方差的方向。该过程中一直重复,重复次数为原始数据中特征的数目。我们会发现,大部分方差都包含在最前面的几个新坐标轴中。因此,我们就可以忽略余下的坐标轴,即对数据进行了降维处理。
2.因子分析(Factor Analysis)。在因子分析中,我们假设在观察数据的生成中有一些观察不到的隐变量(latent variable)。假设观察数据是这些隐变量和某些噪声的线性组合。那么隐变量的数据可能比观察数据的数目少,也就是说通过找到隐变量就可以实现数据的降维。
3.独立成分分析(Independent Component Analysis,ICA)。ICA假设数据是从N个数据源生成的,这一点和因子分析有些类似。假设数据为多个数据源的混合观察结果,这些数据源之间在统计上相互独立的,而在PCA中只假设数据是不相关的。同因子分析一样,如果数据源的数目少于观察数据的数目,则可以实现降维过程。
主成分分析
优点:降低数据的复杂度,识别最重要的多个特征
缺点:不一定需要,且可能损失有用信息
适用数据类型:数值型数据
对于下图中的二维数据,这个二维数据是随机生成的
1 | # coding:utf-8 |
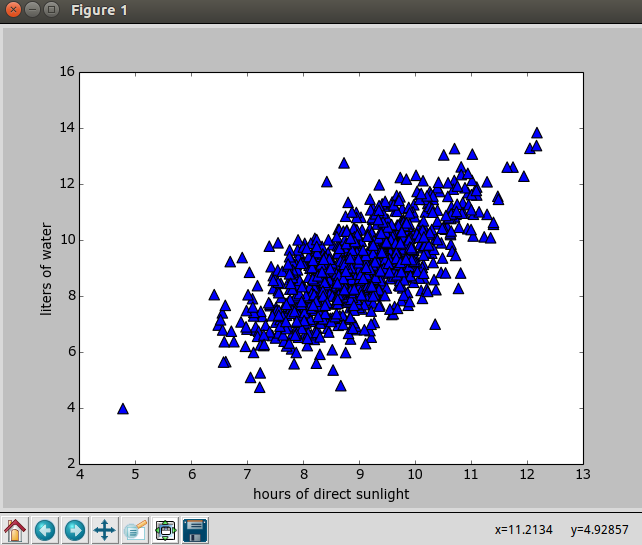
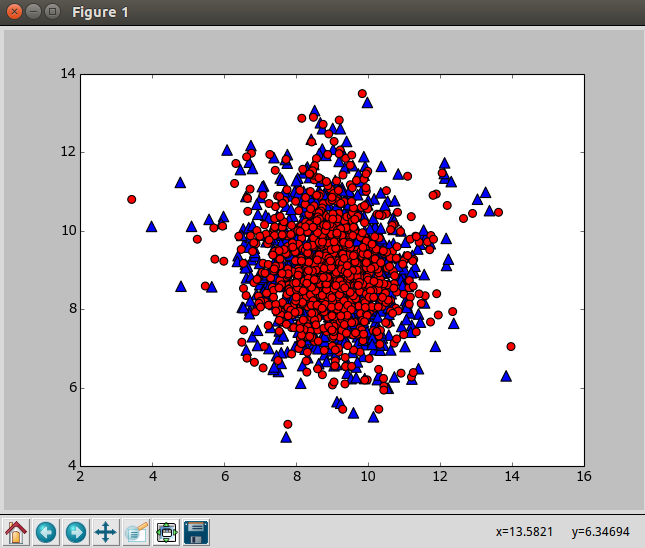
使用下面的程序对二维数据进行主成分分析,输出原始数据重构之后的矩阵lowDDataMat(蓝色),第一主成分reconMat(红色)
1 | def pca(dataMat, topNfeat=9999999): #数据矩阵, 输出前topNfeat个特征,如果topNfeat=1就是降维成1维 |
1 | # coding:utf-8 |
1 | # coding:utf-8 |
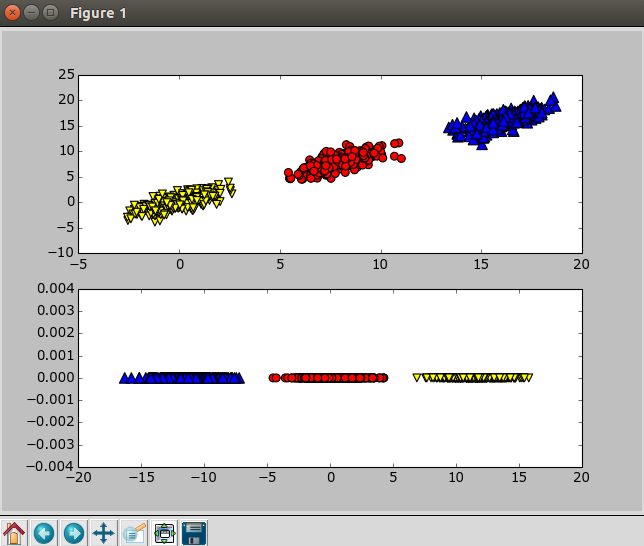
PCA可以从数据中识别其主要特征,它是通过沿着数据最大方差方向旋转坐标轴来实现的。选择方差最大的方向作为第一条坐标轴,后续坐标轴则与前面的坐标轴正交。协方差矩阵上的特征值分析可以用一系列的正交坐标轴来获取。
